

我有一个数据框,其中包含 3 列融化数据(长和细),我想创建一个成对散点图来比较基因(例如。基因 A 与基因 B、基因 A 与基因 C、基因 B 与 C 等)在一个格子中最多 10 个基因。我开始使用 geom_point(),但无法找出在美学中传递 x 和 y 值的最佳方法,所以我开始寻找其他选项,如成对数据,splom、plotmatrix,但我认为数据需要转换为将每个基因作为列和行中的值的格式。
起初这似乎很简单,但我是R/ggplod的新手,似乎无法在网上找到合适的解决方案。
ID Gene value 830 Gene_A 1.8 831 Gene_A 0.4 832 Gene_A 2.5 833 Gene_A 2.3 834 Gene_B -5.1 835 Gene_B 3.6 836 Gene_B 2.0 837 Gene_B 3.2 837 Gene_C -1.6 838 Gene_C -1.4 839 Gene_C -5.5 840 Gene_C -4.4 841 Gene_D -2.7 842 Gene_D -3.2 843 Gene_D -2.5 844 Gene_D -2.5
好的,我可以使用以下代码来解决这个问题:
library(reshape2)
library(ggplot)
#first convert data to generate new ID for each gene
dat$ID<-NULL
dat<-within(dat,{Gene<-as.character(Gene)
ID<- ave(Gene,Gene,FUN=seq_along)
})
请参阅此链接,以获取有关重塑此类数据帧的示例信息。将具有两列的数据帧重塑为具有数据的多列(R)
#cast the new dataset with 1 id per gene, convert from long to wide
dat<-dcast(dat, ID~Gene,value.var="value")
#setup new data frame for plotting
dat4plot=dat
#need to remove ID for plotting
dat4plot$ID=NULL
#Generate plot using pairs function,removed upper panel to see better
pairs(dat4plot,pch=21,bg="red",upper.panel=NULL)
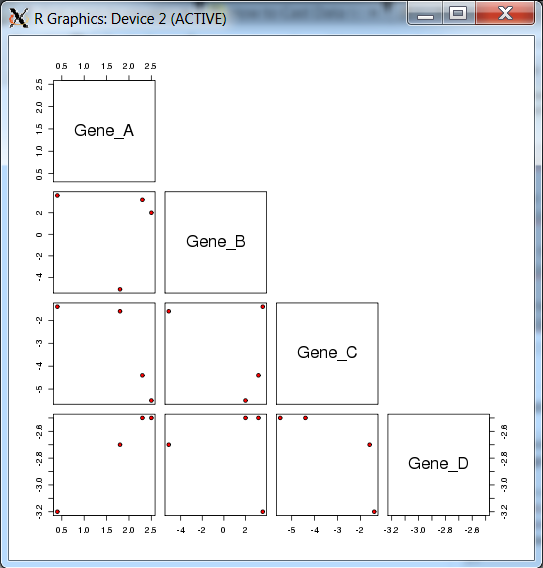
我对此很满意,但也愿意接受更有效的建议!!!